时间:2024-03-13 10:37 来源:未知作者:小芳
3月12日消息,中国科学院自动化研究所多模态人工智能系统实验室杨戈团队与中国科学院生物物理研究所蛋白质科学研究平台生物成像中心孙飞团队合作,以人工智能技术赋能原位结构生物学,提出了一种基于弱监督深度学习的快速准确颗粒挑选方法DeepETPicker。
据悉,相关研究以《DeepETPicker:基于弱监督深度学习的快速准确三维冷冻电子断层扫描图像粒子自动检测器》(DeepETPicker: Fast and accurate 3D particle picking for cryo-electron tomography using weakly supervised deep learning)为题发表于《自然·通讯》(Nature Communications)。
生物大分子(如蛋白质)的结构与功能会随着细胞生理状态的变化不断进行动态调整。原位结构生物学是在接近自然生理状态下研究生物大分子结构和功能的科学,而原位冷冻电镜技术(cryo-electron tomography, Cryo-ET)以其高分辨率和在接近生理条件下观察样品的特点,成为原位结构生物学研究中的关键手段。原位冷冻电镜的技术流程涉及样品制备、数据采集、电子断层重建、颗粒挑选、粒子平均等多个步骤。生物大分子的颗粒挑选,即定位识别,是其中一个关键环节。受限于Cryo-ET图像的极低信噪比和重建伪影等因素,成千上万个目标颗粒的手动挑选极为耗时费力,现有自动挑选方法的应用受到人工标注量高、计算成本高和颗粒质量不理想等多方面限制。
DeepETPicker仅需要少量人工标注颗粒进行训练即可实现快速准确三维颗粒自动挑选。为降低对人工标注量的需求,DeepETPicker优选简化标签来替代真实标签,并采用了更高效的模型架构、更丰富的数据增强技术和重叠分区策略来提升小训练集时模型的性能;为提高颗粒定位的速度,DeepETPicker采用图形处理器(GPU)加速的平均池化-非极大值抑制(MP-NMS, mean pooling and non-maximum suppression)后处理操作,与现有的聚类后处理方法相比提升挑选速度数十倍。此外,为方便用户使用,项目团队推出了操作简洁、界面友好的开源软件(图1)以辅助用户完成图像预处理、颗粒标注、模型训练与推理等操作。
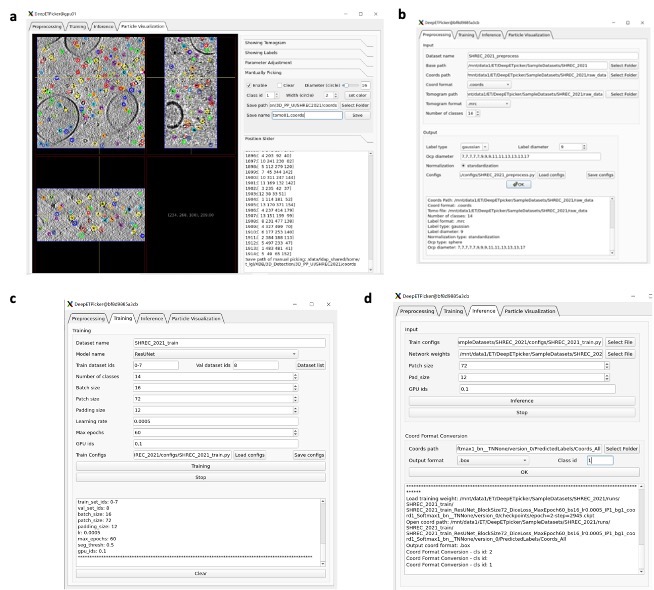
图1 DeepETPicker 软件用户图形界面
使用DeepETPicker从冷冻电子断层扫描图像中挑选颗粒的整体工作流程如图2所示,包括训练阶段(图2a-c)和推理阶段(图2e-g)。在训练数据的准备阶段,研究人员优选了弱标签TBall-M来代替真实掩模以减轻人工标注负担,并在模型架构设计方面,引入坐标卷积(coordinated convolution)和图像金字塔(image pyramid inputs)到3D-ResUNet的分割架构中以提高定位的准确性。在模型推理阶段,DeepETPicker采用重叠断层图分区策略(OT, overlap-tile),避免了由于边缘体素分割精度不佳而产生的负面影响,进而结合MP-NMS操作加速了颗粒中心定位过程。
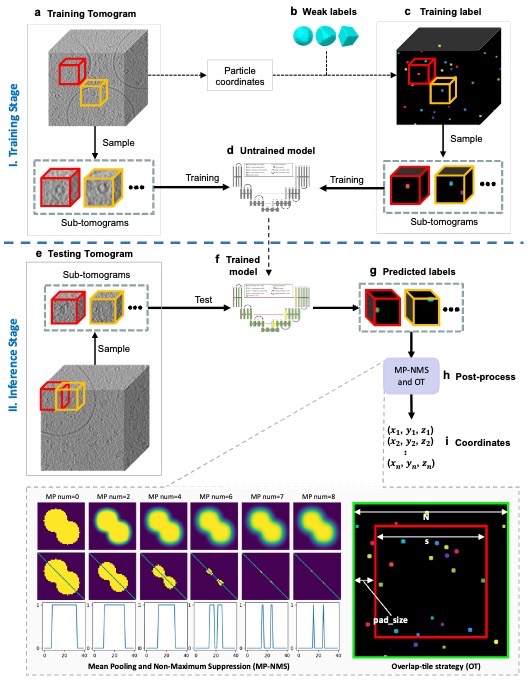
图2 使用 DeepETPicker 从冷冻电子断层扫描图像中挑选颗粒的整体工作流程
研究团队将DeepETPicker与目前性能最优的颗粒挑选方法在多种冷冻电子断层扫描数据集上进行了性能评估对比,采用六个定量指标全面评价颗粒挑选的质量(图3):精确率-召回率(Precision-Recall)、F1-分数(F1-score)、对数似然概率贡献度(Log-likelihood Contribution)、最大值概率(maximum value probability)、RH分辨率(Rosenthal-Henderson resolution)、全局分辨率。结果表明,DeepETPicker在仿真与真实数据集上均可实现快速准确的颗粒挑选,其综合性能明显优于现有的其他方法,生物大分子结构重建达到的分辨率也达到采用专家人工挑选颗粒进行结构重建同样的水平,这进一步体现了DeepETPicker在原位高分辨率结构解析中的实用价值。DeepETPicker将为采用原位冷冻电镜技术的原位结构生物学研究提供有力的支持。
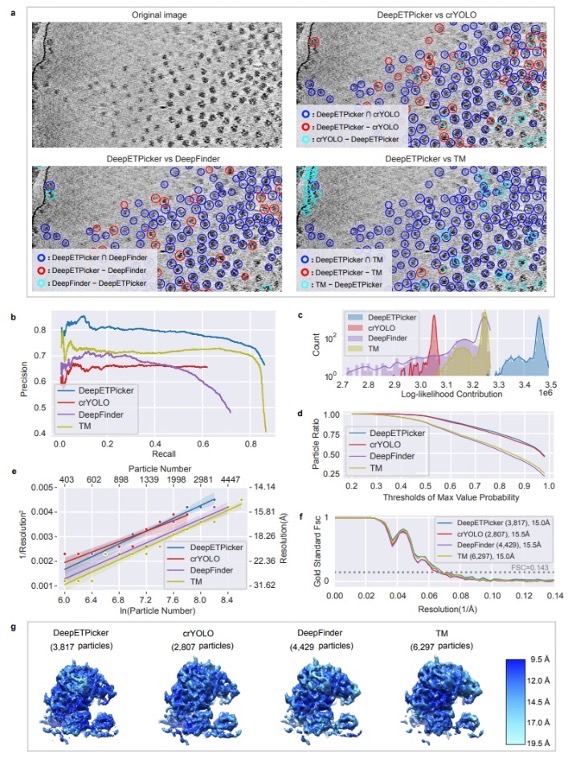
图3 采用定量指标评估DeepETPicker在EMPIAR-10045实验数据集上的粒子挑选性能
更多新闻
相关新闻